Resumen
En el campo de la medicina clínica es crucial poder determinar la seguridad y la eficacia de los fármacos actuales y además acelerar el descubrimiento de nuevos compuestos activos. Para ello se llevan a cabo ensayos de laboratorio, que son métodos muy costosos y que requieren mucho tiempo. Sin embargo, la bioinformática puede facilitar enormemente la investigación clínica para los fines mencionados, ya que proporciona la predicción de la toxicidad de los fármacos y su actividad en enfermedades nuevas, así como la evolución de los compuestos activos descubiertos en ensayos clínicos.
Esto se puede lograr gracias a la disponibilidad de herramientas de bioinformática y métodos de cribado virtual por ordenador (CV) que permitan probar todas las hipótesis necesarias antes de realizar los ensayos clínicos, tales como el docking estructural, mediante el programa BINDSURF.
Sin embargo, la precisión de la mayoría de los métodos de CV se ve muy restringida a causa de las limitaciones presentes en las funciones de afinidad o scoring que describen las interacciones biomoleculares, e incluso hoy en día estas incertidumbres no se conocen completamente. En este trabajo abordamos este problema, proponiendo un nuevo enfoque en el que las redes neuronales se entrenan con información relativa a bases de datos de compuestos conocidos (proteínas diana y fármacos), y se aprovecha después el método para incrementar la precisión de las predicciones de afinidad del método de CV BINDSURF.
Abstract
In the field of clinical research, it is crucial to determine the safety and efficacy of current drugs and further accelerate the discovery of new active compounds. The traditional methods performed expensive laboratory tests. These methods are very costly and require a long time. However, bioinformatics can greatly facilitate the clinical research for the above purposes, providing the prediction of drug toxicity and activity in new diseases and evaluating the validity of active compounds discovered in clinical trials.
This can be achieved through the availability of bioinformatics tools and methods of computer virtual screening (VS) that allow the test all the necessary hypotheses before doing the clinical trials, such as structural docking using the BINDSURF program.
However, the accuracy of most VS methods is severely restricted due to constraints in the affinity or scoring functions describing biomolecular interactions, and even today these uncertainties are not fully known. In this paper we address this problem by proposing a new approach in which neural networks are trained with information on databases of known compounds (target proteins and drugs), for further exploiting the method to improve the accuracy of VS BINDSURF predictions of affinity.
Palabras clave
Redes neuronales ; Investigación clínica ; Descubrimiento de fármacos ; Cribado virtual ; Computación paralela ; Inteligencia computacional
Keywords
Neural networks ; Clinical research ; Drug discovery ; Virtual screening ; Parallel computing ; Computational intelligence
1. Introducción
En la investigación clínica es crucial determinar la seguridad y la eficacia de los fármacos actuales y acelerar el descubrimiento de compuestos activos, especialmente para procesar grandes conjuntos de datos que describen estructuras de proteínas conocidas en bases de datos biológicas, tales como bases de datos de proteínas (PDB, por sus siglas en inglés) [1] , y también derivados de los datos genómicos utilizando técnicas como el modelado de homología [2] . Los ensayos de laboratorio y optimización de compuestos son métodos caros y lentos. Sin embargo, la bioinformática puede facilitar enormemente la investigación clínica para los fines mencionados, proporcionando la predicción de la toxicidad de fármacos y su actividad en enfermedades nuevas, así como la evolución de los compuestos activos descubiertos en ensayos clínicos.
Esto se puede lograr gracias a la disponibilidad de herramientas de bioinformática y métodos de cribado virtual (CV) que permitan probar todas las hipótesis necesarias antes de realizar los ensayos clínicos. Los métodos de CV, como el acoplamiento molecular (docking) , fallan en la predicción de la toxicidad y las predicciones de actividad, ya que están limitados por el acceso a recursos computacionales, e incluso los métodos más rápidos de CV no pueden procesar grandes bases de datos biológicas en un tiempo razonable. Por lo tanto, estas restricciones imponen serias limitaciones en muchas áreas relacionadas de la investigación.
El uso de arquitecturas hardware masivamente paralelas y orientadas al rendimiento, tales como las unidades de procesamiento gráfico (GPU, por sus siglas en inglés), pueden ayudar a superar este problema. Las GPU han ganado popularidad en el campo de la computación de alto rendimiento (HPC, por sus siglas en inglés) mediante la combinación de una impresionante potencia de cálculo con los requisitos más exigentes de gráficos en tiempo real y el lucrativo mercado masivo que supone la industria del videojuego [3] . Los científicos han aprovechado su potencia de cálculo en el dominio computacional y las GPU se han convertido en un recurso clave de aplicaciones en las que el paralelismo es el denominador común [4] . Para mantener este impulso, NVIDIA ha añadido progresivamente nuevas características de hardware para su gama de GPU, con la arquitectura Kepler [5] como hito más reciente. Por lo tanto, las GPU son muy adecuadas para superar la falta de recursos computacionales en los métodos de CV, permitiendo la aceleración de los cálculos necesarios y la introducción de mejoras en los modelos biofísicos no asequibles en el pasado [6] . Hemos trabajado en esta dirección, que muestra cómo los métodos de CV se pueden beneficiar del uso de la GPU [7] , [8] and [9] . Por otra parte, otra carencia importante de los métodos de CV es que por lo general asumen que el lugar de unión que deriva de una sola estructura cristalina será el mismo para los diferentes ligandos, mientras que se ha demostrado que esto no siempre sucede [10] , y por lo tanto es crucial evitar esta simplificación. En este trabajo se presenta una nueva metodología denominada CV BINDSURF que se aprovecha de la alta intensidad de cálculo paralelo masivo de las GPU para acelerar los cálculos requeridos, con máquinas convencionales de bajo consumo y coste, que proporcionan información nueva y útil sobre las proteínas objetivo, con el fin de mejorar las predicciones clave en toxicidad y en grado de actividad. En BINDSURF, una gran base de datos de ligandos se criba simultáneamente sobre toda la superficie de la proteína objetivo. Posteriormente, la información obtenida acerca de los nuevos puntos potenciales de acoplamiento en las proteínas se utiliza para realizar cálculos más detallados utilizando cualquier método de CV, pero solo para un conjunto de ligandos reducido y seleccionado.
Otros autores han realizado estudios de métodos de CV sobre superficies de proteínas enteras [11] utilizando diferentes enfoques y cribando bases de datos de ligandos pequeños pero, por lo que sabemos, ninguno de ellos se ha implementado en GPU ni se ha utilizado de la misma manera que BINDSURF.
Sin embargo, la precisión de la mayoría de los métodos de CV se ve limitada por las limitaciones en las funciones de afinidad o scoring que describen las interacciones biomoleculares, e incluso hoy en día estas incertidumbres no se conocen completamente. En este trabajo abordamos este problema, proponiendo un nuevo enfoque en el que las redes neuronales se entrenan con bases de datos de compuestos activos (fármacos) e inactivos conocidos y posteriormente se utilizan para mejorar las predicciones mediante BINDSURF.
El resto del trabajo se organiza de la siguiente manera. El segundo apartado introduce brevemente el conocimiento previo para comprender mejor el resto del artículo; la tercera parte presenta nuestra propuesta de uso de redes neuronales para mejorar las predicciones de CV; finalmente, se muestran las conclusiones y posibles direcciones para el trabajo futuro.
2. Metodología
En este apartado se describen los métodos que hemos utilizado para la predicción de la afinidad de la proteína-ligando: el método de cribado virtual BINDSURF, una red neuronal entrenada con datos de similitud química de los compuestos activos e inactivos conocidos y la similitud molecular que conforman las variables de entrada de la red neuronal.
2.1. Cribado virtual con BINDSURF
La principal idea que subyace en este método de cribado virtual con BINDSURF es la de una técnica de detección sobre la superficie de proteínas, ejecutada en paralelo sobre GPU. Esencialmente, los métodos de CV procesan una gran base de datos de moléculas con el fin de encontrar cuál encaja mejor con algunos criterios establecidos [12] . En el caso del descubrimiento de nuevos fármacos potenciales, optimización de compuestos, valoración de la toxicidad y las etapas adicionales del proceso de descubrimiento de fármacos, examinamos una gran base de datos de compuestos para encontrar una pequeña molécula, que interactúa de una manera deseada con uno o varios receptores. Entre los muchos métodos de CV disponibles para este propósito hemos decidido utilizar el acoplamiento proteína-ligando [13] and [14] . Estos métodos tratan de obtener predicciones rápidas y exactas de la conformación 3D que adopta un ligando cuando interacciona con una determinada proteína objetivo, y también la fuerza de esta unión, en términos del valor de su función de afinidad. Normalmente, las simulaciones de acoplamiento se llevan a cabo en una parte muy concreta de la superficie de la proteína en métodos como Autodock [15] , Glide [16] y Dock [17] , por nombrar algunos. Esta región se deriva comúnmente desde la posición de un ligando particular en la estructura cristalina, o de la estructura cristalina de la proteína sin ligando. El primero se puede realizar cuando la proteína se ha cocristalizado con el ligando, pero puede ocurrir que no haya una estructura cristalina del par ligando-proteína que se encuentre disponible. Sin embargo, el principal problema es suponer, una vez que se especifica el sitio de unión, que muchos ligandos diferentes van a interactuar con la proteína en la misma región, descartando completamente las otras áreas de la proteína.
Ante este problema, se propone la división de toda la superficie de la proteína en regiones definidas. Posteriormente, se llevan a cabo simulaciones de acoplamiento para cada ligando en todos los puntos especificados de la proteína simultáneamente. Siguiendo este enfoque, los nuevos puntos de acceso se pueden localizar tras un examen de la distribución de la función de afinidad en toda la superficie de la proteína. Esta información podría conducir al descubrimiento de nuevos lugares de unión. Si comparamos este enfoque con una simulación de acoplamiento típico, realizado solo en una región de la superficie, el principal inconveniente de este enfoque radica en el aumento de su coste computacional. Decidimos continuar en esta dirección y mostrar cómo esta limitación puede solucionarse gracias al hardware de las GPU y los nuevos diseños algorítmicos.
En esencia, en una simulación de acoplamiento se calcula la energía de interacción ligando-proteína para una configuración de partida dada del sistema, que está representado por una función de afinidad [18] . En BINDSURF se calcula la función de puntuación, carga electrostática, Van der Waals y los términos de los átomos de hidrógeno (hbond) .
Por otra parte, en los métodos de acoplamiento normalmente se supone que los mínimos de la función de afinidad, entre todas las conformaciones ligando-proteína, representan con exactitud la conformación que adopta el sistema cuando el ligando se une a la proteína [12] . Por lo tanto, cuando se inicia la simulación, tratamos de minimizar el valor de la función de afinidad mediante la introducción continua de perturbaciones aleatorias o predefinidas en el sistema, calculando para cada paso el nuevo valor de la función de afinidad, así como aceptando o no los diferentes enfoques siguiendo el método de reducción de Monte Carlo [19] u otros. Las simulaciones se realizan siempre con un total de 500 pasos de Monte Carlo. Para una discusión detallada es recomendable revisar nuestra anterior publicación BINSURF [20] .
2.2. Redes neuronales
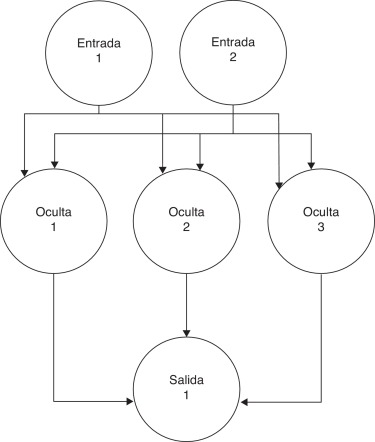
Una de las áreas de aplicación más dominantes de las redes neuronales es la aproximación de funciones no lineales. Hay varios tipos de redes neuronales feedforward ; las que más se utilizan son las redes de múltiples capas con funciones de activación sigmoidal (perceptrones multicapa) y las redes de una sola capa con funciones de activación local (redes de función de base radial). La capacidad de aproximación de las redes neuronales se ha demostrado ampliamente en aplicaciones prácticas y en investigación teórica. Hemos decidido utilizar una red neuronal de una sola capa oculta ( fig. 1 ), ya que se ha probado claramente su impacto en aplicaciones químicas con cálculos similares [21] .
|
|
|
Figura 1. Estructura de la red neuronal con una capa oculta. |
Para tal fin se utilizó la función nnet del paquete R [22] . Se utilizaron por defecto los parámetros que se muestran en la tabla 1 . La elección de estos valores de neuronas de la capa oculta, las iteraciones en la validación cruzada que aseguran la correpresentatividad de todas las muestras y el número de muestras e iteraciones se han validado empíricamente. La conclusión ha sido que los parámetros por defecto son adecuados para los diferentes conjuntos de datos testeados.
| Parámetro | Valor |
|---|---|
| WeighDecayFactor | 0,001 |
| CrossValidationFolds | 5 |
| MaxNumberIterations | 2000 |
| MaxnumberWeights | 2000 |
| TraceOptiimization | No |
2.3. Similitud molecular
Las firmas o huellas (fingerprints) de conectividad extendida (ECFP, por sus siglas en inglés), que se implementan en jCompoundMapper [23] , se utilizaron como descriptores estructurales para la formación de las redes neuronales. Los ECFP son una clase de huellas para la caracterización molecular. Sus características corresponden a la presencia de una estructura exacta (no una subestructura) con puntos especificados de fijación limitados.
En la generación de las huellas, el programa asigna un código inicial para cada átomo. El átomo de código inicial se deriva del número de conexiones con el átomo, el tipo de elemento, la carga y la masa atómicas. Esto corresponde a una ECFP con un tamaño de vecindad cero. Estos códigos átomo se actualizan a continuación, de una manera iterativa, para reflejar los códigos de cada uno de los átomos vecinos. En la siguiente iteración, un esquema de dispersión se emplea para incorporar la información de cada átomo vecino. Para cada nuevo código de átomo se describe entonces una estructura molecular con un tamaño de la vecindad de uno. Este proceso se lleva a cabo para todos los átomos en la molécula. Cuando se alcanza el tamaño de la vecindad que se desea, el proceso se ha completado y el conjunto de todas las características se devuelve como la huella. Para los ECFP empleados en este trabajo se utilizaron tamaños de vecindad de 2, 4 y 6 (ECFP 2, ECFP 4, ECFP 6) para generar las huellas. Los ECFP resultantes pueden representar un conjunto mucho mayor de características que otras huellas y contienen un número significativo de diferentes unidades estructurales cruciales para la comparación molecular entre los compuestos.
3. Resultados y discusión
3.1. Cribado virtual con BINDSURF
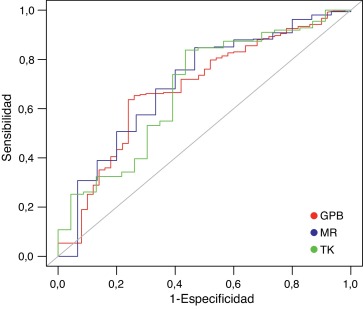
Se realizaron cálculos de CV con BINDSURF usando conjuntos de datos de referencia estándar, como la base de datos directorio de señuelos útiles (DUD, por sus siglas en inglés) [24] , donde los métodos de verificación CV demuestran su eficiencia para diferenciar los ligandos que se sabe que se unen a un objetivo determinado de los no ligantes o señuelos. Los datos de entrada, para cada molécula de cada conjunto, contienen su estructura molecular y muestran si está activo o no. Después de los cálculos con BINDSURF, los resultados para 3 conjuntos distintos de datos DUD se muestran en las curvas ROC de la figura 2 . Teniendo en cuenta los resultados obtenidos para los conjuntos de datos DUD, TK (Thymidine Kinase), MR (Mineralocorticoid Receptor) y GPB (Glycogen Phosphorylase) se caracterizan por el valor del área bajo la curva (AUC, por sus siglas en inglés) para cada curva ROC, y se podría decir que, en promedio, BINDSURF funciona de manera similar a otros métodos para estos conjuntos de datos [25] .
|
|
|
Figura 2. Curvas ROC obtenidas utilizando BINDSURF para los datos DUD, TK (verde), MR (azul) y GPB (rojo). La línea diagonal indica el rendimiento aleatorio. Los valores obtenidos para la AUC son 0,700, 0,695 y 0,675, respectivamente. |
Sin embargo, es evidente que todavía hay margen para la mejora en la función de la afinidad que utiliza BINDSURF y en su método de optimización de la energía (Monte Carlo), que puede afectar directamente a la eficacia de la predicción.
3.2. Predicción de actividad con redes neuronales basada en Similitud molecular
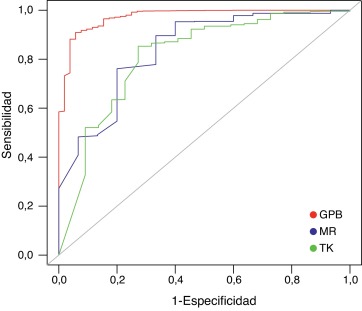
Las redes neuronales fueron entrenadas con los conjuntos de datos DUD mencionados (TK, MR y GPB) y también mediante el uso de las huellas ECFP 2, ECFP 4, ECFP 6, que se calcularon para cada molécula tal como se describe en la sección anterior. Se probaron diferentes combinaciones de estos parámetros (solo ECFP 2, ECFP 2 más ECFP 4, etc.) y se observó que con el uso de forma simultánea de los 3 descriptores se obtuvieron los mejores resultados en términos de AUC para las curvas ROC, como puede verse en la figura 3 ).
|
|
|
Figura 3. Curvas ROC obtenidas utilizando una red neuronal para los datos DUD, TK (verde), MR (azul) y GPB (rojo). La línea diagonal indica el rendimiento aleatorio. Los valores obtenidos para el AUC son 0,801, 0,812 y 0,963, respectivamente. |
Si se comparan estos resultados con los obtenidos anteriormente con BINDSURF (fig. 2 ), los aumentos en la capacidad de predicción son evidentes.
En consecuencia, y teniendo en cuenta la información obtenida por la red neuronal, podemos posprocesar los resultados de acoplamiento obtenidos por la función de afinidad de BINDSURF y rechazar compuestos que se predicen como inactivos. Entonces podemos clasificarlos por el valor final predicho por la función de afinidad para estos casos y estudiar los que visualmente son mejores.
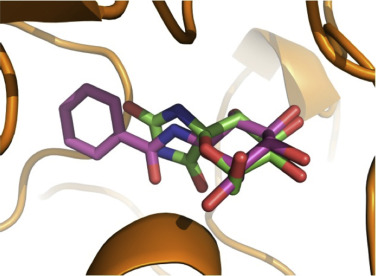
A modo de ejemplo, en la figura 4 se puede observar la coincidencia en la comparación entre la predicción del compuesto de la base de datos DUD para GPB de la parte superior y la pose cristalográfica. En este caso, las principales causas de estabilización se deben a las interacciones de una red de enlaces de hidrógeno, donde el nitrógeno intermedio y los átomos de oxígeno del compuesto predicho caen muy cerca de los mismos átomos de la pose cristalográfica.
|
|
|
Figura 4. Representación del modo de unión encontrado por BINDSURF para el ligando número 17 de los datos DUD conjunto GPB (AP ID: 1A8I) con el esqueleto de color rosa y la pose cristalográfica Beta-D-Glucopiranosa Spirohydantoin, con el esqueleto de color verde. |
4. Conclusiones
En este trabajo se ha mostrado que la capacidad de predicción del método de cribado virtual BINDSURF se puede aumentar usando una red neuronal entrenada con datos de actividad de ligandos. Se debe mencionar que el enfoque de red neuronal solo se puede utilizar cuando hay datos disponibles para los compuestos activos y no activos para una proteína dada.
Esta metodología se puede utilizar para mejorar el descubrimiento de fármacos, su diseño y su reutilización y, por lo tanto, para ayudar considerablemente en la investigación clínica. En próximos trabajos queremos sustituir el algoritmo de minimización de Monte Carlo por alternativas de optimización más eficientes, como el método de optimización de colonia de hormigas, que ya se ha implementado de manera eficiente en GPU [26] y aplicar la flexibilidad total ligando-receptor. Por último, también estamos trabajando en mejorar las funciones de afinidad para incluir de manera eficiente los metales, las interacciones aromáticas y modelos de solvatación.
Agradecimientos
Este trabajo ha sido parcialmente financiado por la Fundación Séneca (Agencia Regional de Ciencia y Tecnología de la Región de Murcia) con beca 18946/JLI/1, del MINECO fondos de la Comisión Europea FEDER en ayudas TIN2009-14475-C04 y TIN2012-31345, así como a la acción Nils Coordinated Mobility con ayuda 012-ABEL-CM-2014A. Agradecemos también a Nvidia Corporation la donación de hardware. También ha sido financiado parcialmente por facilidades del Research Centre for Advanced Technologies (CETA-CIEMAT), con ayuda de European Regional Development Fund (ERDF). CETA-CIEMAT perteneciente CIEMAT y el Gobierno español. Los autores agradecen asimismo los recursos computacionales y el soporte técnico de la Plataforma Andaluza de Bioinformática de la Universidad de Málaga.
Bibliografía
- [1] H.M. Berman, J. Westbrook, Z. Feng, G. Gilliland, T.N. Bhat, H. Weissig, et al.; The protein data bank; Nucleic. Acids. Res., 28 (2000), pp. 235–242
- [2] R. Sánchez, A. Sali; Large-scale protein structure modeling of the Saccharomyces cerevisiae genome ; Proc. Natl. Acad. Sci. U. S. A., 95 (23) (1998), pp. 13597–13602
- [3] M. Garland, D.B. Kirk; Understanding throughput-oriented architectures; Commun. ACM, 53 (2010), pp. 58–66
- [4] M. Garland, S. le Grand, J. Nickolls, J. Anderson, J. Hardwick, S. Morton, et al.; Parallel computing experiences with CUDA; IEEE Micro, 28 (2008), pp. 13–27
- [5] NVIDIA (2012). Whitepaper NVIDIAs Next Generation CUDA Compute Architecture: Kepler GK110.
- [6] H. Pérez-Sánchez, W. Wenzel; Optimization methods for virtual screening on novel computational architectures; Curr. Comput. Aided Drug. Des., 7 (1) (2011), pp. 44–52
- [7] G. Guerrero, H. Pérez-Sánchez, W. Wenzel, J.M. Cecilia, J.M. García; Effective parallelization of non-bonded interactions kernel for virtual screening on gpus. 5th International Conference on Practical Applications of Computational Biology; Bioinformatics (PACBB 2011), Springer Berlin/Heidelberg, 93 (2011), pp. 63–69
- [8] I. Sánchez-Linares, H. Pérez-Sánchez, G.D. Guerrero, J.M. Cecilia, J.M. García; Accelerating multiple target drug screening on gpus. Proceedings of the 9 th International Conference on Computational Methods in Systems Biology (CMSB’ 11); ACM, New York, NY, EE.UU. (2011), pp. 95–102
- [9] I. Sánchez-Linares, H. Pérez-Sánchez, J.M. García; Accelerating grid kernels for virtual screening on graphics processing units; E. D’Hollander, D. Padua (Eds.), Parallel Computing. Proceedings of the International Conference ParCo, 22 (2011), pp. 413–420
- [10] G. Brannigan, D.N. LeBard, J. Henin, R.G. Eckenho, M.L. Klein; Multiple binding sites for the general anesthetic isourane identified in the nicotinic acetylcholine receptor transmembrane domain; Proc. Natl. Acad. Sci. U.S.A., 107 (32) (2010), pp. 14122–14127
- [11] C. Hetényi, D. van der Spoel; Efficient docking of peptides to proteins without prior knowledge of the binding site; Protein. Sci., 11 (7) (2002), pp. 1729–1737
- [12] W. Jorgensen; The many roles of computation in drug discovery; Science, 303 (5665) (2004), pp. 1813–1818
- [13] E. Yuriev, M. Agostino, P.A. Ramsland; Challenges and advances in computational docking: 2009 in review; J. Mol. Recogn., 24 (2) (2011), pp. 149–164
- [14] S.Y. Huang, X. Zou; Advances and challenges in protein-ligand docking; Int. J. Mol. Sci., 11 (8) (2010), pp. 3016–3034
- [15] G. Morris, D. Goodsell, R. Halliday, R. Huey, W. Hart, R. Belew, et al.; Automated docking using a Lamarckian genetic algorithm and an empirical binding free energy function; J. Comput. Chem., 19 (14) (1998), pp. 1639–1662
- [16] R.A. Friesner, J.L. Banks, R.B. Murphy, T.A. Halgren, J.J. Klicic, D.T. Mainz, et al.; Glide: A new approach for rapid, accurate docking and scoring. 1. Method and assessment of docking accuracy; J. Med. Chem., 47 (7) (2004), pp. 1739–1749
- [17] T.J.A. Ewing, S. Makino, A.G. Skillman, I.D. Kuntz; Dock 4.0: Search strategies for automated molecular docking of exible molecule databases; J. Comput.-Aided. Mol. Des., 15 (5) (2001), pp. 411–428
- [18] R. Wang, Y. Lu, X. Fang, S. Wang; An extensive test of 14 scoring functions using the PDBbind refined set of 800 protein-ligand complexes; J. Chem. Inform. Comput. Sci., 44 (6) (2004), pp. 2114–2125
- [19] N. Metropolis, A.W. Rosenbluth, M.N. Rosenbluth, A.H. Teller, E. Teller; Equation of state calculations by fast computing machines; J. Chem. Phys., 21 (1953), pp. 1087–1092
- [20] I. Sánchez-Linares, H. Pérez-Sánchez, J. Cecilia, J. García; High-throughput parallel blind virtual screening using bindsurf; BMC Bioinf., 13 (Suppl 14) (2012), p. S13
- [21] G. Schneider, P. Wrede; Artificial neural networks for computer-based molecular design; Prog. Biophys. Mol. Biol., 70 (3) (1998), pp. 175–222
- [22] W.N. Venables, B.D. Ripley; Modern Applied Statistics with S; (4th ed)Springer, New York (2002)
- [23] G. Hinselmann, L. Rosenbaum, A. Jahn, N. Fechner, A. Zell; jcompoundmap-per: An open source java library and command-line tool for chemical fingerprints; Journal of Cheminformatics, 3 (1) (2011), p. 3
- [24] N. Huang, B.K. Shoichet, J.J. Irwin; Benchmarking sets for molecular docking; J. Med. Chem., 49 (23) (2006), pp. 6789–6801
- [25] J.B. Cross, D.C. Thompson, B.K. Rai, J.C. Baber, K.Y. Fan, Y. Hu, et al.; Comparison of several molecular docking programs: Pose prediction and virtual screening accuracy; J. Chem. Inf. Model., 49 (6) (2009), pp. 1455–1474
- [26] J.M. Cecilia, J.M. García, A. Nisbet, M. Amos, M. Ujaldón; Enhancing data parallelism for ant colony optimization on GPUs; J. Parallel Distr. Com. (2013), pp. 42–51
Document information
Published on 01/12/15
Accepted on 18/06/14
Submitted on 31/01/14
Volume 31, Issue 4, 2015
DOI: 10.1016/j.rimni.2014.06.004
Licence: Other
Share this document
Keywords
claim authorship
Are you one of the authors of this document?