Resumen
La descripción del comportamiento mecánico de tejidos duros mediante el empleo de modelos discretos pasa por diferentes etapas de análisis, desde el procesamiento digital de la imagen hasta la especificación de las propiedades físicas del tejido. Para ello, es necesario tener en cuenta un elemento clave: la descomposición del modelo en sus partes constitutivas. Se realizó un estudio bibliográfico de diversas propuestas para realizar la descomposición y se llegó a la conclusión de la inexistencia de una estrategia única. Existe un cúmulo de propuestas genéricas, pero estas no ofrecen una solución válida a los casos analizados, correspondientes a las articulaciones de la rodilla, la pelvis y el hombro. Por tanto, se propone un algoritmo para realizar la descomposición mediante el análisis de las relaciones espaciales entre los contornos presentes en planos consecutivos, que se basa en 4 etapas: la lectura de los cortes de imágenes de tomografía computarizada; la determinación de los contornos que definen el tejido óseo presente en cada corte; el agrupamiento de los contornos cuya relación espacial cumple un criterio determinado, y la eliminación de los volúmenes no válidos.
Los resultados del algoritmo se compararon con otros obtenidos mediante el empleo de la librería Visualization ToolKit (VTK) y pyFormex, cuyos métodos se utilizan en la visualización y análisis de imágenes médicas y en la modelación de estructuras tridimensionales. Como resultado del algoritmo propuesto tenemos —bajo las mismas condiciones y en un corto tiempo de procesamiento— una descomposición de los modelos anatómicos superior a la realizada por VTK y pyFormex, con aproximadamente el 90% de confianza.
Abstract
Description of mechanical behavior of hard tissues by means of discrete models goes through various stages of analysis, which range from digital image processing to the specification of physical properties of tissue to the discrete model. This requires taking into account a key element: the decomposition of the model into its constituent parts. We conducted a bibliographic study of existing proposals for such decomposition, leading to the conclusion of the absence of a single strategy. There are several generic proposals, but these proved not to give a valid solution applicable to the cases examined corresponding to the articulations of the knee, hip and shoulder. In this paper we propose an algorithm to perform this decomposition by analyzing the spatial relationships between the contours present in consecutive planes. It is based on four stages: reading computer tomography (CT) slices; determining the contours that define bone tissue present on each slice; grouping of contours whose relationship meets a given criterion; and eliminating non-valid volumes.
Results were compared with those obtained by means of Visualization ToolKit (VTK) and pyFormex, widely used in the visualization and analysis of medical imaging and modeling three-dimensional structures. As a main result, proposed algorithm under the same conditions and short processing time performs a better decomposition of anatomical models than the one made by VTK and pyFormex, with about a 90% of confidence.
Palabras clave
Procesamiento de imágenes médicas ; Reconstrucción tridimensional de huesos ; Tomografía computarizada ; Modelación geométrica ; Descomposición
Keywords
Medical image processing ; Bone 3D reconstruction ; CT image ; Geometrical model ; Decomposition
1. Introducción
En la medicina actual, el costo de realizar diagnósticos utilizando imágenes es incalculable. El procesamiento de imágenes de resonancia magnética (RM), tomografía computarizada (TC), tomografía por emisión de positrones, rayos X y otras modalidades, provee un medio no invasivo y efectivo para delinear la anatomía del paciente.
Los modelos anatómicos tridimensionales que se obtienen de tales modalidades reproducen los detalles visuales de las estructuras anatómicas (acelerando la compresión de la forma y la posición espacial), pero distan de ser útiles para el análisis numérico por elementos finitos debido a que carecen de la definición del dominio geométrico de la región anatómica de análisis.
Por este motivo, en el análisis biomecánico se emplean modelos ideales o aproximaciones de las estructuras por estudiar. En consecuencia, debe de existir cierto margen de error entre el comportamiento del modelo generado y el modelo real.
Son varias las aplicaciones médicas en las cuales se utiliza el dominio geométrico, con la premisa de que el error entre el modelo generado y el modelo real sea mínimo, sobre todo en la visualización tridimensional [1] , las mediciones volumétricas [2] and [3] , la cirugía dirigida por imagen [4] , [5] , [6] and [7] y la detección de cambios anatómicos en el tiempo [8] . La descomposición del modelo generado en sus partes constitutivas es un elemento clave y a la vez complejo debido a la proximidad de los huesos en las articulaciones, los efectos de volumen parcial y los factores específicos inherentes a la modalidad de obtención de imágenes [9] .
Realizar la descomposición de forma manual es una tarea ardua, requiere tiempo y no se encuentra exenta de errores a la hora de delimitar las regiones anatómicas (variabilidad intra e interobservador), lo cual demora en gran medida la obtención del dominio geométrico.
Para la automatización del proceso existen varias propuestas, enfocadas en la determinación de los elementos que constituyen una región anatómica específica.
Diversos estudios demuestran la inexistencia de una estrategia única para la separación de los huesos en las articulaciones: Hirano y Hata [10] proponen la aplicación de un sistema experto basado en lógica Fuzzy para la segmentación y la determinación de los huesos y de sus fragmentos en fracturas del pie, considerando para ello 2 tipos de conocimiento: el de la unión y el de la distancia; Mastmeyer et al. [11] introducen una técnica semiautomática para la segmentación y la delimitación de las vértebras en imágenes TC, la cual comienza con la selección manual de la región ocupada por las vértebras bajo la suposición de que estas son cilindros elípticos; los contornos de las vértebras se delimitan mediante la aplicación a los cilindros elípticos de operaciones booleanas y el modelo de balón en el dominio 3D propuesto por [12] and [13] ; Hoad y Martel [7] utilizan el crecimiento de regiones y el filtrado morfológico para segmentar la espina lumbar en imágenes RM.
Por su parte, Sebastian et al. [14] combinan técnicas de contornos activos, de crecimiento y de competición de regiones para la segmentación de los huesos del carpo en imágenes TC; Liu et al. [9] emplean la segmentación livewire para determinar los huesos; Kose et al. [15] utilizan métodos de histograma, segmentación estadística y una plantilla triangular que aplican a la imagen para la detección de los desgarros del menisco en imágenes RM; Zhang et al. [16] proponen el empleo de umbrales adaptativos 3D para la identificación de los huesos; posteriormente, mediante el crecimiento de regiones en 3D se extrae la región específica del hueso; Schmid et al. [17] determinan los huesos presentes en imágenes RM de baja calidad mediante la comparación de modelos ideales de la región de análisis con las imágenes originales.
Dos de las propuestas genéricas existentes para la modelación de estructuras tridimensionales y la separación de estas en sus elementos constitutivos corresponden a criterios de la librería Visualization ToolKit (VTK) y pyFormex.
VTK, desarrollada mediante el concepto de orientación a objetos, se encuentra entre las librerías más populares centradas en la visualización de imágenes médicas, ya que reúne muchas de las características requeridas en la visualización y la gráfica por computadoras, como el procesamiento en paralelo, el renderizado volumétrico, la reconstrucción y el corte de superficies [18] .
pyFormex es un programa de código abierto basado en Python utilizado en la generación, la transformación y la manipulación de modelos geométricos tridimensionales mediante secuencias de operaciones matemáticas. Permite la importación de imágenes de la exploración médica y ofrece una amplia gama de operaciones sobre mallas de superficie; pyFormex también puede ser utilizado como un pre- y posprocesador para los programas de análisis por elementos finitos [19] .
Sin embargo, los resultados obtenidos por esas librerías demostraron que generalmente no se obtiene una correcta separación de los huesos por las articulaciones debido a que carecen, entre otros factores, de elementos para especificar el nivel deseado de descomposición del modelo anatómico.
En cuanto a la utilización de algoritmos de filtrado, si bien ayudan a proporcionar imágenes más nítidas y contrastantes [3] , [20] , [21] , [22] , [23] and [24] , estos procedimientos se fundamentan en transformar y modificar la intensidad de cada píxel. La transformación de intensidad podría eliminar información valiosa o relevante para diagnosticar patologías tempranas.
Por tanto, los autores no incluyeron en la propuesta la utilización de ese tipo de mejoras, aunque sostienen que su aplicación se encuentra en función de la precisión requerida para la descripción geométrica del modelo anatómico.
Se propone un algoritmo para la descomposición de modelos anatómicos tridimensionales en sus partes constitutivas basado en 4 etapas:
- Lectura de los cortes de imágenes TC.
- Determinación de los contornos que definen el tejido óseo en cada corte.
- Formación de los volúmenes, es decir, el agrupamiento de los contornos cuya relación espacial cumple un criterio determinado.
- Eliminación de los volúmenes no válidos.
2. Materiales y método
2.1. Imagen
Se define la imagen en mapa de bits como una matriz que representa la distribución continua de la intensidad de una señal espacial obtenida a intervalos regulares, y la intensidad es cuantificada en un número finito de niveles. Matemáticamente la imagen se define como:
|
|
( 1) |
donde f es la intensidad del píxel y (m, n) definen la posición del mismo a lo largo de un par de vértices ortogonales normalmente definidos como horizontal y vertical. Se asume que la imagen tiene M filas y N columnas, con P niveles discretos de intensidad cuyos valores se encuentran el rango 0 a P–1[25] and [26] .
2.2. Unidades Hounsfield
En las imágenes de TC, la intensidad de la señal espacial está dada por el coeficiente de atenuación lineal μ. Este coeficiente es específico de cada sustancia o materia y expresa la atenuación que sufre un haz de rayos X al atravesar una determinada longitud de una sustancia dada.
La unidad Hounsfield (HU) es una transformación lineal del coeficiente de atenuación lineal μ [27] en que se considera 0 HU la radiodensidad del agua destilada (a presión y temperatura estándar [STP]), mientras que la radiodensidad del aire a STP es –1.000 HU. Cada material posee un coeficiente de atenuación lineal único, y su valor en HU está dado por:
|
|
( 2) |
Aunque varios autores [28] and [29] han calculado los factores de conversión de μ a números HU, estos no han llegado a una solución consistente dado que el valor de μ depende de múltiples factores: intensidad de las radiaciones emitidas, resolución del equipo de adquisición [28] , [30] and [31] y densidad mineral ósea, por lo que no existe un valor único para cada material.
2.3. Umbralización
Es un método de filtrado empleado para etiquetar y agrupar los píxeles cuyas intensidades están en un rango o una función matemática determinada. Generalmente, la umbralización es el paso inicial en una secuencia de operaciones durante el procesamiento de imágenes. Es una de las técnicas más utilizadas, ya que es una de las que más información puede ofrecer, pero al mismo tiempo es bastante subjetiva en su aplicación, puesto que no tiene en cuenta las características espaciales de la imagen y es sensible a inhomogeneidades de la intensidad y al ruido [32] .
Las técnicas de umbralización se dividen en 2 clases: global y local. En la umbralización global se emplea un mismo valor de umbral para toda la imagen, mientras que los métodos de umbralización local calculan un valor de umbral diferente para cada píxel teniendo en cuenta las intensidades de los píxeles que le rodean y una función estadística determinada.
2.4. Imágenes DICOM empleadas
El formato DICOM (Digital Imaging and Communications in Medicine) se emplea para producir, mostrar, almacenar, enviar, procesar, obtener, consultar e imprimir imágenes médicas. Cada imagen obtenida representa un corte de la parte del cuerpo analizada; en estas, cada píxel I = f (m, n) denota el grado de atenuación del haz radiológico sobre el tejido humano. Además de los atributos del píxel, DICOM registra todas las distancias correspondientes, coordenadas 3D y orientaciones, haciendo posible cualquier medición en distancias reales [33] .
Todas las imágenes utilizadas en el presente artículo fueron obtenidas mediante la técnica de TC y corresponden a las regiones anatómicas de la rodilla, el hombro y la pelvis (tabla 1 ).
| Región anatómica | Articulación de la rodilla | Hombro | Pelvis |
| Dispositivo de adquisición | Siemens Sensation 16 | Siemens Sensation 16 | Escáner Philips Mx8000 IDT 16 |
| Energía | 120 kEv, 345 mA | 120 kEv, 238 mA | 140 kEv, 272 mA |
| Dimensiones de la imagen (px) | 512 × 512 × 121 | 512 × 512 × 43 | 512 × 512 × 355 |
| Dimensiones del voxel (mm) | 0,74 × 0,74 × 1,0 | 0,97 × 0,97 × 1,5 | 0,66 × 0,66 × 0,7 |
| Rango del coeficiente de atenuación (HU) | [–1.024,0; 1.607,0] | [–1.024,0; 1.607,0] | [–1.000,0; 1.695,0] |
Por cada región anatómica, se analizaron las imágenes correspondientes a 10 pacientes con idéntica patología y en la misma fase de desarrollo.
2.5. Algoritmo propuesto
El algoritmo propuesto tiene como entrada el conjunto de imágenes DICOM (3) que representan la información correspondiente a la región anatómica de análisis. El intervalo [A, B] es el rango del coeficiente de atenuación obtenido durante la adquisición de la imagen.
|
|
( 3) |
Para clasificar cada píxel como hueso o fondo, se aplica a cada imagen una umbralización global (4), obteniéndose una nueva imagen g formada por los píxeles (m,n) . La selección de (Thglobal) es manual según lo explicado en el epígrafe 2.2.
|
|
( 4) |
La imagen binaria g define el área que ocupa el hueso en la imagen. La descripción geométrica del exterior del hueso (contornos) se define mediante el algoritmo propuesto por Suzuki [34] , obteniéndose por cada imagen segmentada una lista de contornos (5).
|
|
( 5) |
donde: Ck : contorno; R : dimensiones de la imagen R = M · N.
A cada contorno se le asigna una coordenada Z en función de la distancia entre cortes (Dc) y la posición que ocupa la imagen en el conjunto de imágenes de análisis. Para determinar qué contornos pertenecen a una misma isosuperficie, se define el siguiente axioma:
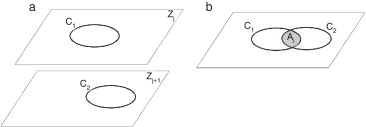
Axioma 1: Dos contornos, en planos consecutivos (fig. 1 a), pertenecen a una misma isosuperficie (estructura anatómica), si se cumple con (6):
|
|
( 6) |
donde Ai es el valor del área de intersección entre estos (fig. 1 b), A1, A2 son las áreas de los contornos y t es el valor permisible. La selección del valor de t está en función de la distancia entre cortes, la región anatómica de análisis y la experiencia adquirida por el especialista.
|
|
|
Figura 1. a) Contornos en planos consecutivos. b) Proyección ortogonal del contorno C1,Zj en el plano Zj+1 y determinación del área de intersección con C2,Zj+1 . |
El proceso consiste en aplicar el axioma 1 a la totalidad de los contornos. Se consideran 3 escenarios posibles: 1) si la relación entre las áreas es mayor o igual a t , entonces (Ck,Zj , Ck,Zj+1 ) forman parte de una misma isosuperficie; en caso contrario, 2) (Ck,Zj , Ck,Zj+1 ) no pertenecen a la misma isosuperficie; 3) si Ck,Zj no tiene relación con ningún contorno del plano Zj+1 , entonces Ck,Zj se considera información no válida y es descartado. Una vez finalizado este proceso, se tiene una lista donde cada elemento representa una isosuperficie, y dentro de esta los contornos que la conforman.
La construcción tridimensional de cada isosuperficie (cerrada y definida por una malla de triángulos) se realiza empleando el algoritmo Marching Cubes[35] . Para una correcta representación espacial se tienen en cuenta los valores de la distancia entre cortes Dc y la distancia entre píxeles Dp (provistos por el formato DICOM).
Si el volumen abarcado por cada isosuperficie construida es inferior a 1,4 mm3 , esta se considera no válida y se descarta. Este volumen se calcula mediante el algoritmo propuesto por [36] , basado en la forma discreta del teorema de la divergencia.
El valor mínimo de 1,4 mm3 se basa en los resultados obtenidos por Bodurka et al. [37] , quienes plantean la existencia de un volumen mínimo para el cual la contribución del ruido fisiológico es igual a la contribución de ruido del sistema de adquisición de imagen.
3. Diseño del experimento
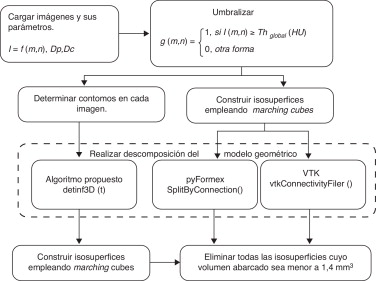
Se desarrollaron 3 aplicaciones (fig. 2 ) para la obtención de estructuras óseas: la primera basada en el algoritmo propuesto en 2.5 y las restantes empleando VTK y pyFormex, respectivamente.
|
|
|
Figura 2. Algoritmos empleados para la definición de estructuras óseas. |
Cada conjunto de imágenes fue procesado indistintamente por las 3 variaciones y los dominios geométricos de las estructuras anatómicas obtenidos fueron comparados empleando como métricas la cantidad de huesos delimitados correctamente y el tiempo de procesamiento.
El hardware utilizado para realizar los experimentos fue un procesador Core I7 2,67 GHz con 4 GB de RAM.
En todos los casos, para clasificar cada píxel como hueso y no hueso, el valor de umbralización global (Thglobal ) se especificó manualmente y se fijó un nivel de confianza del 95%, partiendo de la selección de un intervalo de valores en la escala de Hounsfield, dentro del cual se encuentran los huesos.
Para cada estudio se escogieron los siguientes intervalos: articulación de la rodilla [100; 300], hombro [250; 400] y pelvis fracturada [275; 400]. Al valor óptimo (Thglobal ) se llegó mediante aproximaciones sucesivas hasta obtener una correcta definición del hueso, y en cada aproximación la variación fue de 10 (HU).
De igual forma, el valor permisible de t empleado para la determinación de los contornos que pertenecen a una misma isosuperficie fue especificado manualmente, y se varió entre 0,1 y 0,8 hasta obtener la cantidad máxima de huesos separados correctamente.
4. Resultados y discusión
En el presente trabajo se utilizaron imágenes con bajos niveles de ruido, lo que hizo posible la aplicación de conceptos geométricos para la eliminación de información que no era de interés en lugar de utilizar operaciones de convolución, dado que este tipo de transformaciones podrían eliminar información valiosa o relevante para diagnosticar patologías tempranas.
Las Figura 3 , Figura 4 and Figura 5 muestran un caso de las regiones anatómicas construidas mediante los algoritmos analizados. El nivel de descomposición del dominio geométrico alcanzado por cada una de ellas se observa a través de la representación de las isosuperfices (regiones anatómicas) en diferentes colores.
|
|
|
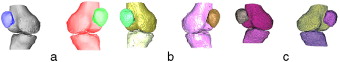
Figura 3. Resultados obtenidos al aplicar los 3 algoritmos a un conjunto de imágenes pertenecientes a la articulación de la rodilla: a) aplicando pyFormex; b) aplicando VTK, y c) aplicando el algoritmo propuesto con t = 0,6. |
|
|
|
Figura 4. Resultados obtenidos al aplicar los 3 algoritmos a un conjunto de imágenes pertenecientes al hombro: a) aplicando pyFormex; b) aplicando VTK, y c) aplicando el algoritmo propuesto con t = 0,2. |
|
|
|
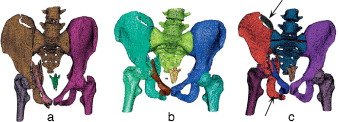
Figura 5. Resultados obtenidos al aplicar los 3 algoritmos a un conjunto de imágenes pertenecientes a una pelvis fracturada: a) aplicando pyFormex; b) aplicando VTK, y c) aplicando el algoritmo propuesto con t = 0,6. |
Los valores de umbralización (Thglobal ) empleados fueron 200 HU, 300 HU y 350 HU para la articulación de la rodilla, el hombro y la pelvis fracturados, respectivamente, confirmándose la imposibilidad de aplicar un valor único para todos los modelos analizados.
En la figura 3 se aprecian las isosuperficies del modelo de la rodilla después de aplicar los 3 algoritmos. pyFormex no separó el fémur de la tibia (fig. 3 a) en ninguna de las 2 piernas, mientras que con la VTK solo se logró separar el fémur y la tibia de la pierna izquierda (fig. 3 b).
El algoritmo propuesto separó satisfactoriamente (fig. 3 c) el fémur de la tibia en ambas piernas. Las rótulas derecha e izquierda fueron identificadas correctamente por los 3 algoritmos.
En la región anatómica del hombro (fig. 4 ) el objetivo fundamental era la obtención por separado de las clavículas, los húmeros y las escápulas. pyFormex identificó correctamente las clavículas pero no separó los húmeros de las escápulas (fig. 4 a). La VTK identificó solamente la clavícula derecha y tampoco separó los húmeros de las clavículas (fig. 4 b).
El algoritmo propuesto identificó correctamente las clavículas y solo separó el húmero izquierdo de la clavícula correspondiente (fig. 4 c).
Finalmente, se utilizaron imágenes de una pelvis fracturada (fig. 5 ). En este caso, pyFormex identificó correctamente el fémur derecho, pero no el izquierdo, al que identificó como parte del hueso iliaco izquierdo. En cuanto al hueso iliaco derecho, lo identificó como parte del sacro (fig. 5 a). La VTK (fig. 5 b) identificó correctamente los huesos iliacos, el fémur derecho y el izquierdo, y el sacro, pero no identificó la fractura de la cresta iliaca izquierda.
El algoritmo propuesto separó los mismos huesos que la VTK, además de identificar correctamente la fractura en la cresta iliaca izquierda como un elemento separado (señalada en la fig. 5 c).
En todos los casos se identificó correctamente la fractura del pilar anterior izquierdo desde el trasfondo acetabular hasta el pubis (señalada en la fig. 5 c). En este tipo de imágenes, el objetivo era determinar las fracturas existentes además de los huesos que constituyen esta región anatómica.
Las Tabla 2 , Tabla 3 and Tabla 4 muestran un análisis general de los resultados obtenidos mediante los algoritmos analizados y registran las estructuras anatómicas que se quería identificar y las que se identificaron correctamente.
| Estructura por identificar/paciente | Algoritmo utilizado | |||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| pyFormex | VTK | Algoritmo propuesto | ||||||||||||||||||||||||||||
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | |
| Fémur derecho | 0 | 1 | 0 | 1 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 1 | 1 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| Fémur izquierdo | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 1 | 1 | 0 | 1 | 0 | 1 | 1 | 1 | 0 | 1 | 1 | 1 | 1 | 0 | 1 | 0 | 1 | 1 | 1 |
| Tibia derecha | 0 | 1 | 0 | 1 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 1 | 1 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| Tibia izquierda | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 1 | 1 | 0 | 1 | 0 | 1 | 1 | 1 | 0 | 1 | 1 | 1 | 1 | 0 | 1 | 0 | 1 | 1 | 1 |
| Rótula derecha | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| Rótula izquierda | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| Estructura por identificar/paciente | Algoritmo utilizado | |||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| pyFormex | VTK | Algoritmo propuesto | ||||||||||||||||||||||||||||
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | |
| Clavícula derecha | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 0 | 0 | 1 | 1 | 1 | 1 |
| Clavícula izquierda | 1 | 1 | 0 | 1 | 0 | 0 | 1 | 1 | 1 | 0 | 1 | 1 | 1 | 1 | 0 | 1 | 1 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| Húmero derecho | 0 | 1 | 1 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 0 | 0 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| Húmero izquierdo | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 1 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| Escápula derecha | 0 | 1 | 1 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 0 | 0 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| Escápula izquierda | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 1 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| Estructura por identificar/paciente | Algoritmo utilizado | |||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| pyFormex | VTK | Algoritmo propuesto | ||||||||||||||||||||||||||||
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | |
| Fémur derecho | 0 | 1 | 1 | 0 | 0 | 1 | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| Fémur izquierdo | 1 | 1 | 1 | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| Hueso ilíaco derecho | 0 | 1 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 0 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 0 |
| Hueso ilíaco izquierdo | 0 | 1 | 0 | 1 | 0 | 0 | 1 | 0 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 0 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 0 |
| Sacro | 0 | 1 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 0 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 0 |
| Fractura | 1 | 0 | 1 | 0 | 1 | 1 | 1 | 0 | 0 | 0 | 1 | 1 | 1 | 0 | 1 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
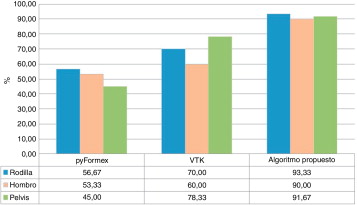
La figura 6 muestra los valores porcentuales obtenidos por cada uno de los algoritmos al determinar correctamente las estructuras anatómicas. Como se puede observar, el algoritmo propuesto obtuvo mayor índice de éxito.
|
|
|
Figura 6. Valores porcentuales obtenidos por cada uno de los algoritmos analizados. |
En la tabla 5 se observa el tiempo de procesamiento promedio empleado por cada algoritmo para la obtención del dominio geométrico de la región anatómica de análisis.
| Región anatómica | Tiempo de procesamiento promedio(s) | ||
|---|---|---|---|
| pyFormex | VTK | Algoritmo propuesto | |
| Articulación de la rodilla | 11,5 | 80,11 | 79,96 |
| Hombro | 6,42 | 33,43 | 44,08 |
| Pelvis | 45,14 | 292,49 | 237,76 |
Según estos resultados, pyFormex es más rápido, pero su utilización implica la no obtención de una correcta descomposición del modelo geométrico. Sin embargo, los tiempos de procesamiento de VTK y el algoritmo propuesto son similares, y este último ofrece mejores resultados en cuanto a la descomposición geométrica.
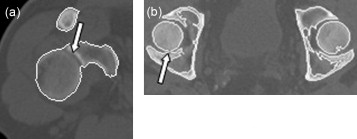
La técnica de umbralización empleada en la determinación de los contornos que definen el tejido óseo en cada corte demostró ser la causa principal por la que no se realiza una correcta descomposición del modelo anatómico. Debido a la pequeña separación de los huesos en las articulaciones, a la posición del paciente al tomar las imágenes y a la limitada resolución espacial de la modalidad de obtención de imágenes empleada, algunos píxeles contienen una mezcla de 2 o más tipos de tejido en lugar de solo uno. Esto se conoce como el efecto de volumen parcial. Los píxeles con este tipo de efecto son difíciles de clasificar con un valor de umbral global.
Como ejemplos, observamos que en la figura 7 a la articulación del hombro derecho se detecta de manera incorrecta, identificándose el húmero como parte de la escápula, y la figura 7 b muestra un corte perteneciente a la pelvis; en este caso, el fémur izquierdo se identifica como parte del hueso iliaco, cosa que no sucede con el lado derecho.
|
|
|
Figura 7. Casos donde falla el algoritmo de detección de contornos: a) articulación del hombro derecho, y b) articulación del fémur. |
5. Conclusiones
En este trabajo se presenta un algoritmo para la descomposición de modelos anatómicos en sus partes constitutivas basado en las relaciones espaciales entre los contornos en planos consecutivos y eliminando la información que no es de interés mediante la utilización de conceptos geométricos.
El algoritmo, bajo las mismas condiciones y en un corto tiempo de procesamiento, define el dominio geométrico de estructuras óseas con resultados superiores a los obtenidos mediante el empleo de VTK y pyFormex, con un nivel de confianza estadístico del 90% para el total de los casos analizados.
El empleo del parámetro t para determinar los contornos que pertenecen a una misma isosuperficie hace posible el control del nivel de partición del modelo anatómico resultante. Además, se concluye que para los modelos anatómicos t debe estar en el rango [0,2; 0,6].
El desempeño del algoritmo propuesto aplicado a estudios de rodilla y pelvis fue superior en comparación con su aplicación a estudios de hombro.
Para una correcta descomposición del modelo anatómico se hace necesario que la técnica de umbralización seleccionada sea capaz de lidiar con el efecto de volumen parcial, haciendo posible la correcta delimitación de las regiones de interés.
Agradecimientos
Los autores agradecen la colaboración prestada durante la elaboración del presente artículo al Dr. Juan Miguel Díaz Quesada y la Lic. Yudith Díaz Gazán.
Bibliografía
- [1] W. Narkbuakaew, S. Sotthivirat, D. Gansawat, P. Yampri, K. Koonsanit, W. Areeprayolkij, et al. 3D surface reconstruction of large medical data using marching cubes in VTK. The 3rd International Symposium on Biomedical Engineering (ISBME 2008), 2008.
- [2] J. Dehmeshki, X. Ye, H. Amin, M. Abaei, X. Lin, S.D. Qanadli; Volumetric quantification of atherosclerotic plaque in CT considering partial volume effect; IEEE Trans. Med. Imaging, 26 (3) (2007), pp. 273–282
- [3] T.M. Nassef; New segmentation approach to extract human mandible bones based on actual computed tomography data; Am. J. Biomed. Eng., 2 (5) (2012), pp. 197–201
- [4] J. Alakuijala; Algorithms for modeling anatomic and target volumes in image-guided neurosurgery and radiotherapy; Med. Phys., 29 (9) (2002), p. 2198
- [5] H. Atmani, F. Merienne, D. Fofi, P. Trouilloud; From medical data to simple virtual mock-up of scapulo-humeral joint; ELCVIA, 7 (3) (2008), pp. 115–128
- [6] S. Sara Mahdavi, N. Chng, I. Spadinger, W.J. Morris, S.E. Salcudean; Semi-automatic segmentation for prostate interventions; Med. Image Anal., 15 (2) (2011), pp. 226–237
- [7] C.L. Hoad, A.L. Martel; Segmentation of MR images for computer assisted surgery of the lumbar spine; Phys. Med. Biol., 47 (2002), pp. 3503–3517
- [8] R.W. McCarley, C.G. Wible, M. Frumin, Y. Hirayasu, J.J. Levitt, I.A. Fischer, et al.; MRI Anatomy of Schizophrenia; Biol. Psychiatry, 45 (1999), pp. 1099–1119
- [9] J. Liu, J.K. Udupa, P.K. Saha, D. Odhner, B.E. Hirsch, S. Seigler, et al.; Rigid model-based 3D segmentation of the bones of joints in MR and CT images for motion analysis; Med. Phys., 35 (8) (2008), pp. 3637–3649
- [10] S. Hirano, Y. Hata; Fuzzy expert system for foot CT image segmentation; Image Vision Comput., 19 (2001), pp. 207–216
- [11] A. Mastmeyer, K. Engelke, C. Fuchs, W.A. Kalender; A hierarchical 3D segmentation method and the definition of vertebral body coordinate systems for QCT of the lumbar spine; Med. Image Anal., 10 (2006), pp. 560–577
- [12] L.D. Cohen, I. Cohen; Finite-element methods for active contour models and balloons for 2-D and 3-D images IEEE Transactions on Pattern Analysis and Machine Intelligence, 15 (11) (1993), pp. 1131–1147
- [13] L.D. Cohen; On active contour models and balloons; CVGIP: Image Understanding, 53 (2) (1991), pp. 211–218
- [14] T.B. Sebastian, H. Tek, J.J. Crisco, B.B. Kimia; Segmentation of carpal bones from CT images using skeletally coupled deformable models; Med. Image Anal., 70 (1) (2003), pp. 21–45
- [15] C. Kose, O. Gengalioglu, U. Sevik; An automatic diagnosis method for the knee meniscus tears in MR images; Expert Syst. Appl., 36 (2009), pp. 1208–1216
- [16] J. Zhang, C.H. Yan, C.K. Chui, S.H. Ong; Fast segmentation of bone in CT images using 3D adaptive thresholding; Comput. Biol. Med., 40 (2010), pp. 231–236
- [17] J. Schmid, J. Kim, N. Magnenat-Thalmann; Robust statistical shape models for MRI bone segmentation in presence of small field of view; Med. Image Anal., 15 (15) (2011), pp. 155–168
- [18] L.S. Avila, S. Barre, M. Geveci; The Visualization ToolKit. Users Guide; Kitware, Inc. (2001)
- [19] Verhegghe B. pyFormex Documentation Release 0.8.9. 2012.
- [20] D. Angelova, L. Mihaylova; Contour segmentation in 2D ultrasound medical images with particle filtering; Mach. Vis. Appl., 22 (3) (2011), pp. 551–561
- [21] T.-C. Lin; Decision-based fuzzy image restoration for noise reduction based on evidence theory; Expert Syst. Appl., 38 (7) (2011), pp. 8303–8310
- [22] H. Liu, C. Yang, N. Pan, E. Song, R. Green; Denoising 3D MR images by the enhanced non-local means filter for Rician noise; Magn. Reson. Imaging, 28 (10) (2010), pp. 1485–1496
- [23] N. Ishak, M. Gangeh, R. Logeswaran, Comparison of denoising techniques applied on low-field MR brain images. En: Fifth International Conference on Computer Graphics, Imaging and Visualization, 2008. CGIV '08. 2008. Penang.
- [24] S. Rueda, J.A. Gil, R. Pichery, M. Alcañiz. Automatic segmentation of jaw tissues in CT using active appearance models and semi-automatic landmarking. En: R. Larsen, M. Nielsen, J. Sporring (Eds.), Medical Image Computing and Computer-Assisted Intervention — MICCAI 2006. Springer Berlin/Heidelberg, 2006, p. 167-174.
- [25] I.N. Bankman; Handbook of Medical Imaging. Processing and Analysis; Academic Press, San Diego, CA (2000)
- [26] A. Flores, A.A. Méndez; Detección estable de los bordes de la oreja en imágenes 2D; Computación y Sistemas, 13 (2) (2009), pp. 195–208
- [27] G.N. Hounsfield; Computed Medical Imaging Nobel Lecture; J Radiol., 61 (6-7) (1980), pp. 459–468
- [28] P. Mah, T.E. Reeves, W.D. McDavid; Deriving Hounsfield units using grey levels in cone beam computed tomography; Dentomaxillofac. Radiol., 39 (2010), pp. 323–335
- [29] M. Lagravère, Y. Fang, J. Carey, R.W. Toogood, G.V. Packota, P.W. Major; Density conversion factor determined using a cone-beam computed tomography unit NewTom QR-DVT 9000; Dentomaxillofac. Radiol., 35 (2006), pp. 407–409
- [30] Molteni R., From CT. Numbers to Hounsfield units in cone beam volumetric imaging: The effect off artifacts. En: 62th AAOMR, 2011, Chicago.
- [31] D. Andisco, S. Blanco, A. Buzzi, S. Ballester; Optimización interdisciplinaria de protocolos en tomografía computada a partir de la modificación del mA y del control del ruido en la imagen; RAR, 74 (4) (2010), pp. 397–402
- [32] Ortega D.R., A.M. Iznaga. Técnicas de segmentación de imágenes médicas. En: 14 Convención Científica de Ingeniería y Arquitectura. Ciudad de La Habana, 2008.
- [33] NEMA, Digital Imaging and Communications in Medicine (DICOM). 2011, National Electrical Manufacturers Association, 1300 N. 17th Street. Rosslyn, Virginia 22209 USA.
- [34] S. Suzuki, K. Abe; Topological structural analysis of digitized binary images by border following; Comput. Vis. Graph. Image Process., 30 (1) (1985), pp. 32–46
- [35] W.E. Lorensen, H.E. Cline; Marching cubes: A high resolution 3D surface construction algorithm; ACM SIGGRAPH Comput. Graphics, 21 (4) (1987), pp. 164–169
- [36] A.M. Alyassin, J.L. Lancaster, J.H. Downs 3rd, P.T. Fox; Evaluation of new algorithms for the interactive measurement of surface area and volume; Med. Phys., 21 (6) (1994), pp. 741–752
- [37] J. Bodurka, F. Ye, N. Petridou, K. Murphy, P.A. Bandettini; Mapping the MRI Voxel Volume in Which Thermal Noise Matches Physiological Noise-Implications for fMRI; Neuroimage, 34 (2) (2007), pp. 542–549
Document information
Published on 06/11/13
Volume 29, Issue 4, 2013
DOI: 10.1016/j.rimni.2013.11.001
Licence: Other
Share this document
Keywords
claim authorship
Are you one of the authors of this document?