Resumen
La descripción del comportamiento mecánico de tejidos duros mediante el empleo de modelos discretos pasa por diferentes etapas de análisis, que van desde el procesamiento digital de la imagen y la especificación de las propiedades físicas del tejido hasta el modelo discreto.
Las recientes tecnologías de adquisición de imágenes médicas permiten su obtención con elevada resolución. La obtención del dominio geométrico de estas requiere de elevados niveles de hardware para su procesamiento y almacenamiento, y la simplificación poligonal, con un error mínimo definido, es una de las vías para la compresión del dominio geométrico. La importancia de la simplificación de polígonos es la de contar con la información suficiente y necesaria para la reconstrucción de superficies y volúmenes que describan geométricamente órganos y tejidos del cuerpo humano, para su posterior análisis mediante elementos finitos.
En el presente trabajo se determina de forma experimental un parámetro de control para la simplificación de los polígonos presentes en imágenes médicas.
Los resultados experimentales demostraron que es posible la obtención de un modelo tridimensional con una variación volumétrica promedio del 87,49% empleando el 36,3% de los datos iniciales, con un error máximo del 1,0% entre las áreas inicial y final de los polígonos presentes, en un menor tiempo de procesamiento.
Abstract
The description of mechanical behavior of hard tissues by means of discrete models goes through various stages of analysis, which range from digital image processing and the specification of physical properties of tissue to the discrete model.
Nowadays, technologies for acquiring medical images allow obtaining high resolution images. Obtaining the geometrical domain of these images requires high levels of hardware to process and store them, and polygonal simplification, with a defined minimum error, is one of the ways to compress the geometrical domain. Polygonal simplification is important in order to have the necessary and sufficient information to reconstruct surfaces and volumes that geometrically describe tissues and organs of the human body for their analysis by means of finite elements.
In this paper an experimental control parameter is defined for polygon simplification.
Experimental results proved that is possible to obtain a tridimensional model with an average volume variation of 87.49%, using only 36.3% of initial data, with a maximum error of 1.0% between initial and final polygon areas. All this process takes less processing time.
Palabras clave
Optimización poligonal ; Compresión de datos ; Imagen médica
Keywords
Polygonal optimization ; Data compression ; Medical image
1. Introducción
El volumen de información que se obtiene como resultado de la transformación de las imágenes que representan una estructura anatómica en un modelo tridimensional para estudios biomédicos es elevado. Su almacenamiento, análisis matemático y representación gráfica son tareas complejas.
Se considera que la simplificación de la información comienza por el análisis de los puntos hasta llegar a las estructuras más complejas, como las superficies y los volúmenes. La propuesta de estudio se centra en el dominio bidimensional, por lo cual la estructura geométrica por simplificar son los polígonos. Mediante la aplicación de estas transformaciones en imágenes capturadas por equipos de escaneo, el objetivo del proceso es simplificar la base de datos manteniendo las propiedades topológicas.
El problema puede enunciarse de la siguiente forma: aproximar el polígono Q (con m puntos) a un polígono P (con n puntos), tal que m < n , con una medida de error menor que un valor dado y con la premisa de que se mantengan los puntos donde se localizan los máximos locales de curvatura absoluta y las propiedades geométricas del polígono (posición espacial, orientación, área y perímetro).
Este problema aparece como un subproblema en sistemas de información geográfica, cartográfica, gráfica por computadoras, imágenes médicas y compresión de datos. Las recientes tecnologías de adquisición de imágenes médicas y satelitales permiten la obtención de imágenes de elevada resolución y su análisis requiere un elevado tiempo de procesamiento. Por tanto, los algoritmos de simplificación se emplean para simplificar y comprimir estas imágenes para un almacenamiento o una transmisión eficientes [1] .
Se define como curva poligonal cerrada (polígono) a la región del plano limitada por una polilínea cerrada, formada por un conjunto ordenado de vértices: P = {p1 , p2 , .., pn } = {(x1 , y1 ), (x2 , y2 ), …, (xn , yn )}, que cumplen con algunas propiedades [2] .
En el campo de la simplificación poligonal se comenzó con la determinación de los puntos dominantes en las curvas digitales. El algoritmo Rosenfeld-Johnston [3] fue uno de los primeros, y permitió la obtención de los puntos dominantes según un radio de vecindad de 1/10 a 1/15 de los puntos iniciales, tomando como parámetro de control el radio de curvatura. Su aplicación fundamental fue el reconocimiento de caracteres.
Teh y Chin [4] proponen la automatización de la selección del radio de vecindad y mantienen el radio de curvatura como variable por minimizar mediante un procedimiento que cubre 3 etapas. Trabajos más recientes en esta dirección presentan una técnica denominada Partición Multinivel de la Unidad (Partition Multi-level of Unity , MPU) para la creación de superficies suaves y cerradas a partir de un conjunto de contornos 2D que han sido extraídos de un escaneo 3D [5] . Se basa en el ajuste de los puntos del contorno a un B-Spline cuadrático, cuyos parámetros se calculan a partir del gradiente de los contornos en la imagen. El error de aproximación se controla mediante el cálculo de la distancia de Taubin [6] .
Otra tendencia en la simplificación de polígonos es obtener curvas simplificadas a partir de considerarlas segmentos de recta. El algoritmo Ramer-Douglas-Peucker tiene como propósito, a partir de una curva compuesta por segmentos de rectas, encontrar la curva simplificada y suave consistente en un subconjunto de los puntos que definían la curva original [7] and [8] . Es un algoritmo recursivo cuyo parámetro de control lo constituye la desviación: distancia mínima entre el punto de análisis y el segmento de recta que se está analizando.
En cuanto a los valores óptimos para la optimización poligonal, en un estudio Casadei y Mitter asumen la distancia mínima para que 2 puntos pertenezcan a una misma curva de 0,75 unidades píxel [9] . Más tarde, Kolesnikov y Fränti plantean que la aproximación de curvas poligonales con un mínimo de error (problema min-ɛ) puede ser resuelta mediante el empleo de la programación dinámica o mediante la teoría de grafos [10] and [11] .
Respecto al estudio de las propiedades geométricas de los polígonos y los aspectos que tener en cuenta para comparar si 2 polígonos son similares, Veltkamp plantea que una secuencia infinita de momentos p, q = 0,1,… , determina la forma de un objeto de forma única y viceversa. Por tanto, es posible definir un conjunto de funciones invariantes que representan diferentes aspectos de los contornos [12] . Las funciones invariantes empleadas las plantea Hu [13] , quien demostró que los momentos de los contornos son invariantes a la traslación, la rotación y el cambio de escala, excepto por el séptimo valor cuyo signo lo cambia la reflexión.
Trabajos recientes aplicados en el campo del procesamiento de imágenes médicas utilizan la simplificación poligonal para optimizar la representación de imágenes vectoriales obtenidas [14] , [15] , [16] , [17] and [18] .
En cuanto a la simplificación de isosuperficies, son varias las propuestas. Schmitt et al. [19] describen un método recursivo de subdivisión adaptativa que aproxima una nube de puntos a una superficie de parches bicúbica Bernstein-Bézier. Como restricción, un parche debe ser continuo con sus parches vecinos. Se comienza por una aproximación burda de la superficie; si esta no se encuentra dentro de la tolerancia definida por el usuario, cada parche es subdivido en parches más pequeños. El proceso finaliza cuando el conjunto de parches que se aproxima a los datos de entrada se encuentra dentro de la tolerancia especificada.
De Haemer y Zyda [20] desarrollaron una variación del método anterior empleando parches planos rectangulares; Schroeder et al. [21] proponen un método denominado triangle decimation que reduce el número de caras de una malla triangular en un porcentaje especificado mediante la eliminación iterativa de vértices; Hamann [22] describe un método que emplea la eliminación iterativa de las caras triangulares ordenadas mediante estimados de la curvatura y de la forma; Turk [23] propone introducir nuevos vértices en la representación original en las localizaciones de máxima curvatura; posteriormente se construye una nueva triangulación que incluye los vértices originales y los nuevos y, mediante un operador de reducción local, se eliminan los puntos originales; Hoppe et al. [24] desarrollaron un algoritmo de optimización de mallas mediante un esquema de minimización de la energía, y Kalvin y Taylor [25] presentan un algoritmo que simplifica mallas poliédricas dentro de tolerancias especificadas previamente. Basado en un criterio de aproximación de bordes, el algoritmo une adaptativamente los polígonos coplanares redundantes, preservando la forma del poliedro original.
Para la construcción de isosuperficies tridimensionales de modelos anatómicos es necesaria la determinación de los contornos, presentes en cada corte, que definen la estructura anatómica de interés.
Los métodos basados en la detección de contornos permiten la delimitación de fronteras entre regiones, caracterizadas por puntos de alto valor de gradiente [26] . Estos se aplican en imágenes de características bien definidas, con amplio contraste, aunque también pueden producir resultados no satisfactorios debido a su sensibilidad al ruido.
En 1985, Suzuki y Abe [27] presentan 2 algoritmos para el análisis topológico de imágenes binarias digitales. El primero determina las relaciones entre los contornos exteriores e interiores de una imagen binaria. En el segundo algoritmo, que es una versión modificada del primero, solo se detectan los contornos exteriores. Ambos algoritmos se basan en las relaciones de conectividad entre los píxeles presentes en la imagen, que constituye la primera propuesta para la obtención de los contornos en una imagen binaria.
El algoritmo de Suzuki realiza un escaneo sobre una imagen binaria. Cuando encuentra un píxel, analiza si este pertenece a un borde o si es un borde exterior y le asigna un número único (NBD), el cual se actualiza en función de las relaciones de conectividad del píxel con sus vecinos. Al mismo tiempo se actualiza un árbol que contiene la información de las relaciones entre los contornos.
El crecimiento de regiones es un procedimiento mediante el cual se agrupan píxeles o subregiones en regiones mayores [28] . El procedimiento más sencillo se denomina unión de píxeles: comienza a partir de un conjunto de píxeles semilla que crece añadiendo píxeles a dicha semilla de entre aquellos píxeles vecinos que tienen propiedades similares (nivel de gris, relaciones de conectividad, etc.). El procedimiento termina cuando no existen más píxeles en el vecindario de la región que cumplan el criterio de similitud. Se toma como nueva semilla otro píxel que no esté incluido en alguna de las regiones ya determinadas y el proceso termina cuando todos los píxeles de la imagen pertenezcan a una región determinada [29] , [30] , [31] and [32] .
Los métodos de segmentación basados en contornos activos son capaces de interpretar rasgos dispersos e integrarlos para obtener la superficie de los objetos de interés. Se definen como curvas elásticas que evolucionan alrededor del objeto a segmentar [33] , y los más conocidos son Snakes y Level sets[33] and [34] .
Si bien se han reportado resultados satisfactorios en su aplicación, se trata de esquemas computacionalmente intensivos y plantean algunos inconvenientes, según se describe en [35] , principalmente cuando los objetos por segmentar poseen formas irregulares.
El algoritmo Level sets es uno de los más utilizados en la segmentación de imágenes. Debido a que es capaz de ajustarse automáticamente a los cambios topológicos, posee una elevada estabilidad numérica e independencia de la parametrización, pero su principal desventaja es que incrementa la complejidad computacional [36] . Entre los trabajos más recientes se pueden mencionar [36] , [37] , [38] , [39] and [40] .
La importancia de la simplificación de polígonos es poder contar con la información suficiente y necesaria para la construcción de superficies y volúmenes que describan geométricamente órganos y tejidos del cuerpo humano para su análisis mediante elementos finitos.
La principal desventaja de los métodos de simplificación espacial existentes radica en que exigen la construcción de la superficie como en [21] , [22] , [23] , [24] and [25] o el análisis completo de la nube de puntos como plantean [19] and [20] , lo cual demanda un tiempo adicional de procesamiento.
En tal sentido, se hace la propuesta de simplificar la información en el plano. Dicha simplificación, aplicada a los polígonos, utiliza un parámetro de control para garantizar la variación mínima del área.
2. Materiales y método
2.1. Imagen
Se define la imagen en mapa de bits como una matriz que representa la distribución continua de la intensidad de una señal espacial obtenida a intervalos regulares, y la intensidad se cuantifica en un número finito de niveles. Matemáticamente, la imagen se define como:
|
|
( 1) |
donde f es la intensidad del píxel y (m, n ) definen su posición a lo largo de un par de vértices ortogonales usualmente definidos como horizontal y vertical. Se asume que la imagen tiene M filas y N columnas, con P niveles discretos de intensidad cuyos valores se encuentran el rango 0 a P –1 [41] and [42] .
2.2. Imágenes DICOM empleadas
El formato DICOM (Digital Imaging and Communications in Medicine) se emplea para producir, mostrar, almacenar, enviar, procesar, obtener, consultar e imprimir imágenes médicas. Cada imagen obtenida representa un corte de la parte del cuerpo analizada; cada píxel I = f (m, n) denota el grado de atenuación del haz radiológico sobre el tejido humano. Además de los atributos del píxel, DICOM registra todas las distancias correspondientes, coordenadas 3D y orientaciones, haciendo posible cualquier medición en distancias reales [43] .
Las imágenes utilizadas en el presente artículo fueron obtenidas mediante la técnica de tomografía computarizada, y se corresponden con las regiones anatómicas del fémur, pies, hombro y tibia-peroné.
- Fémur: 208 cortes de 355 × 251 píxeles con un tamaño de vóxel de 0,68 × 0,68 × 1,0 mm3 .
- Pies: 250 cortes de 512 × 512 píxeles con un tamaño de vóxel de 0,74 × 0,74 × 1,0 mm3 .
- Hombro: 43 cortes de 512 × 512 píxeles con un tamaño de vóxel de 0,97 × 0,97 × 1,0 mm3 .
- Pierna: 121 cortes de 512 × 512 píxeles con un tamaño de vóxel de 0,97 × 0,97 × 1,0 mm3 .
2.3. Existencia del parámetro de control t
Para simplificar los vértices de un polígono manteniendo la variación del área con un error mínimo se propone que la definición de polígono se exprese como los puntos que definen su contorno o perímetro y el área que estos encierran:
|
|
( 2) |
Tomando como fundamento este principio se considera que un polígono Q es la simplificación de un polígono P , si la diferencia entre las áreas de P y Q es como máximo el 1,0% del área de P :
|
|
( 3) |
Como algoritmo de simplificación se propone utilizar el de Douglas-Peucker. Este se basa en el principio de que una polilínea puede ser descrita como un segmento de recta si la distancia entre el segmento de recta (SR) que une puntos extremos de la polilínea y su punto más alejado de SR es menor que un valor ɛ dado [8] .
Se plantea la existencia de una relación entre ɛ, la variación del área que encierra el polígono y su cantidad de puntos. Esta relación se define como el parámetro de control t y para su determinación se propone el algoritmo 1.
Algoritmo 1. Algoritmo para la determinación del valor de tolerancia t en función de la variación del área
| Entrada: Perímetro C | |
| (1) | AINICIAL = área del polígono dado por el perímetro C |
| (2) | PINICIAL , PFINAL = cantidad de puntos de C |
| (3) | t = 0,01 |
| (4) | mientras ( ) y (PFINAL > 4) |
| (5) | C1 = simplificar_perímetro (C, t) |
| (6) | AFINAL = área del polígono dado por el perímetro C1 |
| (7) | PFINAL = cantidad de puntos de C1 |
| (8) | t = t + 0,01 |
| Salida: AINICIAL , PINICIAL , AFINAL , PFINAL , t | |
En la simplificación de polígonos mediante Douglas-Peucker la secuencia de puntos por analizar la definen los puntos extremos del polígono: el primer punto (E1 ) y el punto (E2 ) más alejado de E1 en el sentido de orientación del polígono. De la secuencia {E1 ,…, E2 } se determina el punto A, cuya distancia d al segmento de recta es la mayor de la secuencia y se compara con la tolerancia t , con las siguientes posibilidades:
Si d ≤ t , todos los puntos situados entre se eliminan.
Si d > t , el algoritmo se aplica recursivamente en 2 nuevas polilíneas y .
Si no existen puntos entre , la polilínea no es reducible.
t se convertirá en el parámetro de control para diferentes tipos de polígonos definidos en la imagen y se tendrá que comprobar si al aplicarlo a diferentes estudios médicos se obtiene una simplificación del volumen de datos.
3. Determinación experimental del parámetro de control t
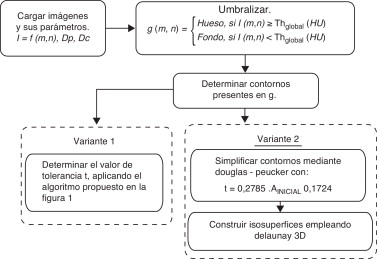
Los contornos que definen las estructuras óseas del fémur y de los pies se obtuvieron mediante el algoritmo propuesto en la figura 1 , variante 1. El algoritmo propuesto tiene como entrada un conjunto de imágenes DICOM que representan la región anatómica de análisis. El intervalo [A, B] es el rango del coeficiente de atenuación (HU).
|
|
( 4) |
En cada imagen, los píxeles se clasifican como como hueso o fondo mediante la umbralización global, y se obtiene una nueva imagen g .
|
|
( 5) |
La descripción geométrica del exterior de cada hueso (contornos) se define mediante el algoritmo propuesto por Suzuki y Abe [27] . Por cada imagen segmentada se obtiene una lista de contornos.
|
|
( 6) |
|
|
|
Figura 1. Algoritmos utilizados. Variante 1, utilizada para la determinación estadística del parámetro de control t . Variante 2, utilizada en la simplificación y reconstrucción del modelo tridimensional. |
Para determinar la relación entre ɛ, la variación del área que encierra el polígono y su cantidad de puntos, se simplificó cada contorno de Lc mediante el algoritmo 1, y como datos de análisis se obtuvieron: área, perímetro, cantidad de puntos inicial y final, t y porcentaje de compresión.
El valor de compresión establece la relación entre la cantidad de vértices inicial (PINICIAL ) y final (PFINAL ) de un polígono una vez simplificado.
|
|
( 7) |
En total, se analizaron 4.084 polígonos (tabla 1 ).
| Variable | Mínimo | Máximo |
|---|---|---|
| Área (px)2 | 1,5 | 5.373 |
| Cantidad de puntos | 5 | 452 |
| Perímetro (px) | 6,82 | 996,24 |
Se observó que al aplicar el algoritmo de simplificación (algoritmo 1) a polígonos cuya cantidad de puntos era menor que 4 o el área encerrada por estos inferior a 1,0 px2 , el resultado era un segmento de recta, lo cual no se considera válido en el estudio de modelos biomédicos. En tal sentido, los polígonos que cumplían algunas de las características expresadas anteriormente se consideraron simplificados y no formaron parte de la muestra estadística.
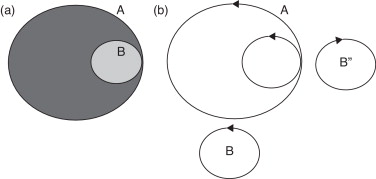
En el caso de los dominios múltiplemente conexos, en la figura 2 a se observa la imagen en mapa de bits de una circunferencia A, a la que es tangente interior una circunferencia B. El algoritmo de segmentación empleado dará como resultado los polígonos A y B orientados en el sentido antihorario y el polígono B” orientado en sentido horario, el cual es un hueco de A (fig. 2 b). Por ello, fue posible el análisis de todos los polígonos presentes, independientemente de las posiciones relativas entre estos.
|
|
|
Figura 2. Análisis de superficies con hueco: a) imagen en mapa de bits, b) resultado de la segmentación. |
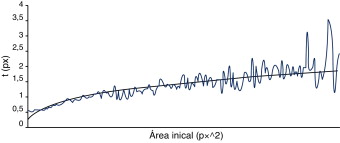
Con los datos obtenidos, se analizó la relación entre el área inicial, el perímetro inicial y la cantidad de puntos inicial con respecto a t . Como resultado se observó una mayor correlación de datos entre área inicial y t .
Para un mejor análisis de la correlación entre el área inicial vs. t , las áreas iniciales se agruparon en intervalos de 25 px2 (fig. 3 ), calculándose para cada intervalo la t promedio. Un análisis de las diferentes ecuaciones de tendencia demostró que la función de potencia fue la que mejor se ajustó a los datos.
|
|
( 8) |
|
|
|
Figura 3. Comportamiento del área inicial vs. t . |
En cuanto al nivel de compresión, fue posible disminuir la cantidad de vértices al 39,34% con una variación máxima del área del 1%.
4. Resultados
Para validar la ecuación de simplificación, se aplicó el algoritmo propuesto en la figura 1 (variante 2) a un estudio del hombro. Inicialmente, la malla (fig. 4 a) contenía 27.274 vértices y 64.359 triángulos, y la reconstrucción del modelo aplicando la triangulación de Delaunay tardó 11 segundos. La figura 4 b muestra el modelo simplificado, en el que se obtienen 9.748 vértices y 23.363 triángulos, con un tiempo de reconstrucción de 1 segundo. La compresión de datos fue del 35,74%, con una variación volumétrica del 17,18% (tabla 2 ).
|
|
|
Figura 4. Estudio del hombro: a) isosuperficie no simplificada, b) isosuperficie simplificada con la ecuación propuesta. |
| Modelo simplificado | Vértices | Triángulos | Volumen (px3 ) | Tiempo de reconstrucción (s) | ||||
|---|---|---|---|---|---|---|---|---|
| Total | % del original | Total | % del original | Valor | % del original | Valor | % del original | |
| Hombro | 9.748 | 35,74 | 23.363 | 36,30 | 308.659,08 | 82,82 | 1 | 9,09 |
| Pierna | 15.429 | 36,86 | 26.847 | 40,27 | 338.572,47 | 92,16 | 2 | 13,33 |
Los resultados obtenidos (tabla 2 ) al aplicar el algoritmo propuesto en la figura 1 (variante 2) en un estudio de la pierna fueron: el modelo simplificado (fig. 5 b) se reconstruyó en apenas 2 segundos, constaba de 15.429 vértices (36,86% de los valores iniciales) y 26.847 triángulos (40,27% de los valores iniciales), para una compresión del 36,86% de los datos, y con una variación volumétrica del 7,84%.
|
|
|
Figura 5. Estudio de la pierna. a) isosuperficie no simplificada, b) isosuperficie simplificada con la ecuación propuesta. |
5. Conclusiones
Se demostró que el valor de simplificación t se encuentra en función del área inicial del polígono y presenta un comportamiento de función de potencia.
Como promedio, con el 36,3% de los datos iniciales fue posible la obtención de isosuperficies con una variación volumétrica del 87,49%.
En cuanto al tiempo de procesamiento, el modelo tridimensional se reconstruyó utilizando como promedio el 11,21% del tiempo que se necesitaría para reconstruirlo con la totalidad de los puntos.
La probabilidad de simplificación de un polígono con área mayor a 69 px2 es del 77,5%, y el 100% de los polígonos con área mayor a 69 px2 se pueden simplificar.
Considerando como información no válida en la imagen médica aquellos polígonos con área menor a 1,5 mm2 , y teniendo en cuenta la resolución que esta representa en la realidad, se observó que los contornos que cumplen esta condición se encontraban fundamentalmente en el grupo de los que no se pudieron simplificar.
Como trabajo futuro, teniendo en cuenta las 2 últimas conclusiones, sería posible incluir reglas al algoritmo de simplificación para que este decida si es efectivo simplificar un polígono dado. Esto podría disminuir aún más el tiempo necesario para la obtención del dominio geométrico de un modelo anatómico.
Agradecimientos
Los autores agradecen la colaboración prestada durante la elaboración del presente artículo al Dr. Juan Miguel Díaz Quesada y la Lic. Yudith Díaz Gazán.
Bibliografía
- [1] O. Daescu, N. Mi; Polygonal chain approximation: A query based approach; J. Comput. Geom, 30 (2005), pp. 41–58
- [2] A.M. Iznaga Benítez, I. Pérez Mallea; Fundamentos de la gráfica por computadoras; Editorial Félix Varela (2006)
- [3] A. Rosenfeld, E. Johnston; Angle detection on digital curves; IEEE Trans. Comput., 22 (1973), pp. 875–878
- [4] C.H. Teh, R.T. Chin; On the detection of dominant points on digital curves; IEEE Tr. PAMI, 11 (8) (1989), pp. 859–872
- [5] I. Braude, J. Marker, K. Museth, J. Nissanov, D. Breen; Contour-based surface reconstruction using MPU implicit models; Graphical Models, 69 (2) (2007), pp. 139–157
- [6] G. Taubin; Estimation of planar curves, surfaces, and nonplanar space curves defined by implicit equations with applications to edge and range image segmentation; IEEE Trans. Pattern Anal. Mach. Intell., 13 (11) (1991), pp. 1115–1138
- [7] U. Ramer; An iterative procedure for the polygonal approximation of plane curves; Comput. Gr Image Process, 1 (3) (1972), pp. 244–256
- [8] D. Douglas, T. Peucker; Algorithms for the reduction of the number of points required to represent a digitized line or its caricature; Cartographica, 10 (2) (1973), pp. 112–122
- [9] S. Casadei, S. Mitter; Hierarchical Image Segmentation—Part I: Detection of Regular Curves in a Vector Graph; Int. J. Comput. Vision, 27 (1) (1998), pp. 71–100
- [10] A. Kolesnikov, P. Fränti; Reduced-search dynamic programming for approximation of polygonal curves; Pattern Recognit. Lett., 24 (14) (2003), pp. 2243–2254
- [11] P.-F. Marteau, G. Ménier; Speeding up simplification of polygonal curves using nested approximations; Pattern Anal Appl (2008), pp. 1–8
- [12] Veltkamp RC (2001). Shape matching: Similarity measures and algorithms in International Conference on Shape Modeling and Applications. Genova; Italy; p. 188-197.
- [13] M. Hu; Visual pattern recognition by moment invariants; IEEE Trans. Inf. Theory, 8 (2) (1962), pp. 179–187
- [14] F. Lecron, S.A. Mahmoudi, M. Benjelloun, S. Mahmoudi, P. Manneback; Heterogeneous computing for vertebra detection and segmentation in X-ray images; Int. J. Biomed. Imaging (2011) ID 640208
- [15] W.L. Nowinski, I. Volkau, Y. Marchenko, A. Thirunavuukarasuu, T.T. Ng, V.M. Runge; A 3D model of human cerebrovasculature derived from 3T magnetic resonance angiography; Neuroinformatics, 7 (1) (2009), pp. 23–36
- [16] W.F. Sensakovic, A. Starkey, S.G. Armato; A modified gradient correlation filter for image segmentation: Application to airway and bowel; Med. Phys., 32 (2) (2009), pp. 480–485
- [17] W.L. Nowinskia, B.C. Chua, Y. Marchenko, F. Puspitsari, I. Volkau, M.V. Kno; Three-dimensional reference and stereotactic atlas of human cerebrovasculature from 7 Tesla; NeuroImage, 55 (3) (2011), pp. 986–998
- [18] D. Waanders, D. Janssen, M. Berahmani, M.A. Miller, K.A. Mann, N. Verdonschot; Interface micromechanics of transverse sections from retrieved cemented hip reconstructions: An experimental and finite element comparison; J. Mater. Sci. Mater. Med., 23 (8) (2012), pp. 2023–2035
- [19] F.J.M. Schmitt, E. Barsky, W.-H. Du; An adaptive subdivision method for surface-fitting from sampled data; Comput. Graphics (Proc. Siggraph), 20 (4) (1986), pp. 179–188
- [20] M.J. DeHaemer, M.J. Zyda; Simplification of objects rendered by polygonal approximations; Comput. Graphics, 15 (2) (1991), pp. 175–184
- [21] W.J. Schroeder, J.A. Zarge, W.E. Lorensen; Decimation of triangle meshes; Comput. Graphics (Proc. Siggraph), 26 (2) (1992), pp. 65–70
- [22] E. Hamann; A data reduction scheme for triangulated surfaces; Comput. Aided Geom, 11 (2) (1994), pp. 197–214
- [23] G. Turk; Re-tiling of polygonal surfaces; Comput. Graphics (Proc. Siggraph), 26 (2) (1992), pp. 55–64
- [24] H. Hoppe, T. DeRose, T. Duchamp, J. McDonald, W. Stuetzle; Mesh Optimization; Proceedings of SIGGRAPH, ACM Press, Anaheim, CA (1993)
- [25] A.D. Kalvin, R.H. Taylor; Superfaces: Polygonal Mesh Simplification with Bounded Error; IEEE Comput. Graphics Appl. (1996)
- [26] A.K. Jain; Fundamentals of Digital Image Processing; Prentice Hall (1989)
- [27] S. Suzuki, K. Abe; Topological structural analysis of digitized binary images by border following; Comput. Vis.raph. Image Process, 30 (1) (1985), pp. 32–46
- [28] S.W. Zucker; Region growing: Chilhood and adolescence; Comp. Graphics Image. Proc., 5 (1976), pp. 382–399
- [29] J. Alakuijala; Algorithms for modeling anatomic and target volumes in image-guided neurosurgery and radiotherapy; Med. Phys., 29 (9) (2002), p. 2198
- [30] M.C.d. Andrade; An interactive algorithm for image smoothing and segmentation; ELCVIA, 4 (2) (2004), pp. 32–48
- [31] A.v. Wangenheim, R.F. Bertoldi, D.D. Abdala, M.M. Richter, L. Priese, F. Schmitt; Fast two-step segmentation of natural color scenes using hierarchical region-growing and a color-gradient network; J. Braz Comp. Soc., 14 (2008), pp. 29–40
- [32] D. Angelova, L. Mihaylova; Contour segmentation in 2D ultrasound medical images with particle filtering; Mach. Vis. Appl., 22 (3) (2011), pp. 551–561
- [33] M. Kass, A. Witkin, D. Terzopoulos; Snakes: Active contour models; Int. J. Comput. Vision, 1 (4) (1988), pp. 321–331
- [34] V. Caselles, R. Kimmel, G. Sapiro; Geodesic Active Contours; Int. J. Comput. Vision, 22 (1) (1997), pp. 61–79
- [35] L. Ji, H. Yan; Loop-free snakes for highly irregular object shapes; Pattern Recognit. Lett., 23 (2002), pp. 579–591
- [36] J. Gai, R.L. Stevenson; Robust contour tracking based on a coupling between geodesic active contours and conditional random fields; J. Visual Commun. Image Represent., 22 (1) (2011), pp. 33–47
- [37] Z. Ma, R.N. Jorge, T. Mascarenhas, J.M.R.S. Tavares; Segmentation of female pelvic cavity in axial T2-weighted MR images towards the 3D reconstruction; Int. J. Numer. Methods Biomed. Eng., 28 (6-7) (2012), pp. 714–726
- [38] Z. Ma, R.N. Jorge, T. Mascarenhas, J.M.R.S. Tavares; Novel approach to segment the inner and outer boundaries of the bladder wall in T2-weighted magnetic resonance images; Ann. Biomed. Eng., 39 (8) (2011), pp. 2287–2297
- [39] Z. Ma, R.N. Jorge, T. Mascarenhas, J.M.R.S. Tavares; A shape guided C-V model to segment the levator ani muscle in axial magnetic resonance images; Med. Eng. Phys., 32 (7) (2010), pp. 766–774
- [40] K.C. Ciesielski, J.K. Udupa; A framework for comparing different image segmentation methods and its use in studying equivalences between level set and fuzzy connectedness frameworks; Comput. Vision Image Understanding, 115 (6) (2011), pp. 3305–3318
- [41] I.N. Bankman; Handbook of Medical Imaging. Processing and analysis; Academic Press (2000)
- [42] A. Flores, A.A. Méndez; Detección estable de los bordes de la oreja en imágenes 2D; Comp. y Sist., 13 (2) (2009), pp. 195–208
- [43] NEMA, Digital Imaging and Communications in Medicine (DICOM), 2011, National Electrical Manufacturers Association, 1300N. 17th Street. Rosslyn, Virginia 22209 USA.
Document information
Published on 01/03/15
Accepted on 04/12/13
Submitted on 17/05/13
Volume 31, Issue 1, 2015
DOI: 10.1016/j.rimni.2013.12.003
Licence: Other
Share this document
Keywords
claim authorship
Are you one of the authors of this document?